Laboratorio 4 - Parte 2. Regularización de modelos.#
!wget -nc --no-cache -O init.py -q https://raw.githubusercontent.com/jdariasl/Intro_ML_2025/master/init.py
import init; init.init(force_download=False); init.get_weblink()
from local.lib.rlxmoocapi import submit, session
import inspect
session.LoginSequence(endpoint=init.endpoint, course_id=init.course_id, lab_id="L04.02", varname="student");
#configuración del laboratorio
from Labs.commons.utils.lab4 import *
_ = part_2()
import seaborn as sns
En este laboratorio vamos analizar el efecto del sobre-ajuste (over-fitting), como identificarlo y como podemos regualizar los modelos para evitarlo o disminuir su efecto.
En este laboratorio, vamos a enfocarnos en 2 modelos (usando la librería de sklearn):
Regresión logística
MLP
El sobre-ajuste también puede ser causado por la maldición de la dimensionalidad. No vamos enfocarnos en como tratar esta condición ya que esto lo vamos a ver un poco más adelante cuando evaluemos las técnicas de selección de características.
Vamos usar la base de datos iris para realizar nuestra practica. Vamos a convertir el problema a un problema de clasificación biclase
x,y = load_wine(return_X_y=True)
unique, counts = np.unique(y, return_counts=True)
print("distribución original (claves de las etiquetas, valores de número de muestras): \n", dict(zip(unique, counts )))
y = np.where(y==0, 0, 1)
unique, counts = np.unique(y, return_counts=True)
print("distribución luego de conversión (claves de las etiquetas, valores de número de muestras): \n", dict(zip(unique, counts )))
Una de las condiciones para que se presenten sobre-ajustes es tener un conjunto de entrenamiento pequeño.
En nuestra practica vamos a simular esta condición para ver que técnicas podemos usar para reducir el efecto del sobre-ajuste.
Nota
En un problema real, si se observa que las medidas de rendimiento no satisfacen las necesidades, la respuesta puede ser que se necesiten más datos en el conjunto de entrenamiento. Las condiciones que usaremos en esta práctica son para ver el efecto del sobre ajuste.
# simular conjunto de datos pequeño
x, x_test, y, y_test = train_test_split(x, y, test_size=0.6, random_state=10, stratify = y)
scaler = StandardScaler().fit(x)
x = scaler.transform(x)
x_test = scaler.transform(x_test)
Ejercicio 1 - Detectar sobre ajuste#
En nuestro primer ejercicio vamos a crear una función para detectar las diferencias entre los errores de entrenamiento y de prueba.
Calcular error de entrenamiento y prueba
La función recibe de manera arbitraria un estimador de sklearn
Se debe retornar la diferencia absoluta (solo numeros positivos) entre entrenamiento y prueba.
# ejercicio de código
def diff_train_test(Xtrain, Ytrain, Xtest, Ytest, sklearnModel):
"""función que retorna error de entrenamiento
Xtrain: matriz numpy con las muestras de entrenaniento
Ytrain: matrix numpy con las etiquetas de entrenamiento
Xtest: matriz numpy con las muestras de prueba
Ytest: matrix numpy con las etiquetas de prueba
sklearnModel: objeto estimador de sklearn ya entrenado
retorna: tupla con tres elementos:
error entrenamiento, error test y
diff absoluta entre error y test
"""
error_train =
error_test =
diff =
return (error_train, error_test, diff)
Registra tu solución en línea
student.submit_task(namespace=globals(), task_id='T1');
Con la función construida, vamos a usarla para verificar la differencia entre el error de entrenamiento y prueba para los dos modelos que vamos a usar:
MLP con dos capas, cada una con 64 neuornas.
random_state=1es usado para lograr tener los mismos resultados siempreRegresión logistica forzada para que no use ninguna regularización.
random_state=1es usado para lograr tener los mismos resultados
mlp = MLPClassifier(hidden_layer_sizes=[20,20], max_iter=500, alpha =1e-6, random_state=1)
mlp.fit(x, y)
# aca usamos el * para pasa cadar elemento como argumento
print("MLP entrenamiento:{0:.3f}, test:{1:.3f} y diff {2:.3f}".format(*diff_train_test(x,y, x_test, y_test,mlp)))
reg = LogisticRegression(penalty=None, max_iter=500, random_state=1)
reg.fit(x, y)
print("Logistic Regresion entrenamiento:{0:.3f}, test:{1:.3f} y diff {2:.3f}".format(*diff_train_test(x,y, x_test, y_test, reg)))
Ejercicio 2 - Experimentar con MLP regularizado#
Vamos a comenzar a regularizar el modelo, el primer metodo que usaremos es el de parada anticipada (early-stopping). Este ya se encuentra implementado dentro de la libreria, vamos a experimentar con este parametro y el número de neuronas en el MLP.
#@title Pregunta Abierta
#@markdown ¿Explique en sus palabras a que corresponde el metodo de parada anticipada?
respuesta = "" #@param {type:"string"}
#@title Pregunta Abierta
#@markdown ¿Basandose en la documentación de sklearn para MLPClassifier que relación tiene el parametro validation_fraction con la parada anticipada?
respuesta = "" #@param {type:"string"}
# ejercicio de código
def exp_mlp_early_stop(num_neurons, is_early_stop, Xtrain,Xtest,Ytrain, Ytest):
""" función para realizar experimentos con el MLP y early stopping
num_neurons: list de enteros con el número de neuronas a usar
is_early_stop: list de boolean para confirmar si se aplica early stop
Xtrain: matriz de numpy con las muestras de entrenamiento
Xtest: matriz de numpy con las muestras de prueba
ytrain: vector numpy con las etiquetas de entrenamiento
ytest: vector numpy con las etiquetas de prueba
Retorna: dataframe con 5 columnas:
- número de neuronas
- error de entrenamiento
- error de prueba
- diferencia entrenamiento y prueba
- flag early stopping
"""
resultados = pd.DataFrame()
idx = 0
for early_stop in is_early_stop:
for neurons in num_neurons:
#Haga el llamado a la función para crear y entrenar el modelo usando los datos de entrenamiento
# prestar atención a los parametros, correctos.
hidden_layer_sizes = tuple(2*[neurons])
# llame el parametro para que el MLP pare anticipadamente
# Ajuste la semilla en 1 para garantizar la reproducibilidad de los resultados
mlp = MLPClassifier(hidden_layer_sizes= hidden_layer_sizes, max_iter = 1000,...)
# entrenar
mlp.
# llamar a la funcion creada anteriomente
error_train, error_test, diff = ...
resultados.loc[idx,'neuronas en capas ocultas'] = neurons
resultados.loc[idx,'error de entrenamiento'] = ...
resultados.loc[idx,'error de prueba'] = ...
resultados.loc[idx,'diferencia entrenamiento y prueba'] = ...
resultados.loc[idx,'is_early_stop'] = early_stop
idx+=1
return (resultados)
Registra tu solución en línea
student.submit_task(namespace=globals(), task_id='T2');
res_early_stop = exp_mlp_early_stop( [4,8,16], [True, False], x, x_test, y, y_test)
sns.relplot(x = 'neuronas en capas ocultas', y='diferencia entrenamiento y prueba', hue = 'is_early_stop', data = res_early_stop, kind = 'line', aspect=2)
plt.show()
Ejercicio 3: Regularización L2#
#@title Pregunta Abierta
#@markdown ¿Explique en sus palabras en qué consiste la regularización L2?
respuesta = "" #@param {type:"string"}
# ejercicio de código
def exp_mlp_l2(num_neurons, l2_values, Xtrain,Xtest,Ytrain, Ytest):
""" función para realizar experimentos con el MLP con regularización L2
num_neurons: list de enteros con el numero de neuronas a usar
l2: list de floats con valores para regularizacion l2
Xtrain: matriz de numpy con las muestras de entrenamiento
Xtest: matriz de numpy con las muestras de prueba
ytrain: vector numpy con las etiquetas de entrenamiento
ytest: vector numpy con las etiquetas de prueba
Retorna: dataframe con 5 columnas:
- número de neuronas
- error de entrenamiento
- error de prueba
- diferencia entrenamiento y prueba
- valor de l2
"""
resultados = pd.DataFrame()
idx = 0
for l2 in l2_values:
for neurons in num_neurons:
#Haga el llamado a la función para crear y entrenar el modelo usando los datos de entrenamiento
# prestar atención a los parametros, correctos.
hidden_layer_sizes = tuple(2*[neurons])
# llame el parametro adecuado del MLPClassifier
# Ajuste la semilla en 1 para garantizar la reproducibilidad de los resultados
mlp = MLPClassifier(hidden_layer_sizes= hidden_layer_sizes, max_iter = 1000, ...)
mlp
# llamar la funcion creada anteriomente
error_train, error_test, diff = ...
resultados.loc[idx,'neuronas en capas ocultas'] = neurons
resultados.loc[idx,'error de entrenamiento'] = ...
resultados.loc[idx,'error de prueba'] = ...
resultados.loc[idx,'diferencia entrenamiento y prueba'] = ...
resultados.loc[idx,'l2'] = l2
idx+=1
return (resultados)
Registra tu solución en línea
student.submit_task(namespace=globals(), task_id='T3');
res_l2 = exp_mlp_l2([4,16,64], [1e-3,1e-1,1e0, 1e1, 2e2, 1e3], x, x_test, y, y_test)
sns.relplot(x = 'l2', y='diferencia entrenamiento y prueba',
hue = 'neuronas en capas ocultas',
data = res_l2, kind = 'line',
aspect=2, palette=sns.color_palette('viridis', n_colors=res_l2['neuronas en capas ocultas'].nunique()))
plt.show()
Ejercicio 4 - Experimentar con regresión logistica regularizada#
Ahora vamos explorar la opciones de regularización de la regresión logistica. En la libreria se implementan más formas de regularizar, pero solo vamos a comprobar la regularización de norma L2.
# ejercicio de código
def exp_reg_l2(l2_values, Xtrain,Xtest,Ytrain, Ytest):
""" función para realizar experimentos con el LR con regularización L2
l2_values: list de floats con valores para regularizacion l2
Xtrain: matriz de numpy con las muestras de entrada
Xtest: matriz de numpy con las muestras de prueba
ytrain: vector numpy con las etiquetas de las muestras de entrenamiento
ytest: vector numpy con las etiquetas de las muestras de prueba
Retorna: dataframe con 4 columnas:
- Valor del parámetro de regularización
- error de entrenamiento
- error de prueba
- diferencia entre error de entrenamiento y prueba
"""
resultados = pd.DataFrame()
idx = 0
for l2 in l2_values:
#Defina y entrene el modelo LR usando los datos de entrenamiento
# prestar atención a los parametros, correctos. Para lograr
# la regularizacion deseada (pasar el valor de "l2" directamente al
# parámetro de la libreria asociado)
# Ajuste la semilla a 1 para garantizar la reproducibilidad de los resultados
reg = LogisticRegression(max_iter = 500, ...)
# llamar la funcion creada anteriomente
error_train, error_test, diff = ...
resultados.loc[idx,'l2'] = l2
resultados.loc[idx,'error de entrenamiento'] = ...
resultados.loc[idx,'error de prueba'] = ...
resultados.loc[idx,'diferencia entrenamiento y prueba'] = ...
idx+=1
return (resultados)
Registra tu solución en línea
student.submit_task(namespace=globals(), task_id='T4');
reg_l2 = exp_reg_l2([1e-6,1e-3,1e-1,1e0, 1e1], x, x_test, y, y_test)
g = sns.relplot(x = 'l2', y='diferencia entrenamiento y prueba',
data = reg_l2, kind = 'line',
aspect=2)
g.set(xscale="log")
plt.show()
#@title Pregunta Abierta
#@markdown ¿Qué efecto tiene el parametro que controla L2 en la regresión logistica en relación con el sobreajuste? ¿Es diferente al MLP?
respuesta= "" #@param {type:"string"}
Ejercicio 5 Efecto del tamaño del conjunto de entrenamiento#
Finalmente como mencionamos anteriormente, en los ejercicios que hemos resuelto estábamos simulando la situación de un conjunto de datos de entrenamiento pequeño. En nuestro último ejercicio vamos a comprobar el efecto del tamaño del conjunto de entrenamiento.
# ejercicio de codigo
def train_size_experiments(train_pcts,X,Y,sk_estimator):
"""función que realiza experimentos para
comprobar la influencia del tamaño del conjunto
de entrenamiento.
train_pcts: lista de floats con los % de entrenamiento a evaluar
X: matriz de numpy con el conjunto de muestras
Y: vector numpy con las etiquetas de salida correspondientes
sk_estimator: estimador/modelo de sklearn definido (sin entrenar)
Retorna: dataframe con 4 columnas:
- tamaño del conjunto de entrenamiento
- error de entrenamiento
- error de prueba
- diferencia entrenamiento y prueba
"""
resultados = pd.DataFrame()
idx = 0
for train_pct in train_pcts:
#complete con el parámetro train_pct (recuerde que son proporciones)
Xtrain, Xtest, Ytrain, Ytest = train_test_split(X, Y, stratify = Y, random_state=10, ...)
# normalizamos
scaler = StandardScaler().fit(Xtrain)
Xtrain = scaler.transform(Xtrain)
Xtest = scaler.transform(Xtest)
# entrenar!
sk_estimator.fit(X=..., y=...)
# llamar la funcion creada anteriomente
error_train, error_test, diff = diff_train_test(...)
resultados.loc[idx,'error de entrenamiento'] = ...
resultados.loc[idx,'error de prueba'] = ...
resultados.loc[idx,'diferencia entrenamiento y prueba'] = ...
# complete con el tamaño del entrenamiento
resultados.loc[idx,'tamaño de entrenamiento'] = ...
idx+=1
return (resultados)
Registra tu solución en línea
student.submit_task(namespace=globals(), task_id='T5');
x,y = load_iris(return_X_y=True)
unique, counts = np.unique(y, return_counts=True)
print("distribución original (claves las etiquetas, valores el número de muestras): \n", dict(zip(unique, counts )))
y = np.where(y==2, 0, 1)
unique, counts = np.unique(y, return_counts=True)
print("distribución luego de conversión (claves las etiquetas, valores el número de muestras): \n", dict(zip(unique, counts )))
# comprobamos con un MLP
mlp = MLPClassifier(hidden_layer_sizes=[64,64], max_iter=1000, random_state=1)
train_size_exp = train_size_experiments([0.2,0.3,0.5,0.7,0.9], x, y, mlp)
# vemos las tres medidas
ax = train_size_exp.plot(x="tamaño de entrenamiento", y="error de entrenamiento", color="b", legend=False, figsize = (9,6))
train_size_exp.plot(x="tamaño de entrenamiento", y="error de prueba", ax=ax, legend=False, color="r")
ax2 = ax.twinx()
ax2.set_ylabel("diff train y test")
ax.set_ylabel("eficiencia")
train_size_exp.plot(x="tamaño de entrenamiento", y="diferencia entrenamiento y prueba", ax=ax2, legend=False, color="k")
ax.figure.legend()
plt.show()
Notas Finales
Para tener en cuenta: Sklearn tiene una función que realiza algo similar a lo que creamos en el anterior ejercicio y que usamos en la clase sobre complejidad de modelos.
Debemos notar que en esta practica exageramos algunas situaciones para lograr medir y ver el efecto del sobre-ajuste. En un problema fuera de la didactica del laboratorio un flujo de trabajo ideal es el siguiente:
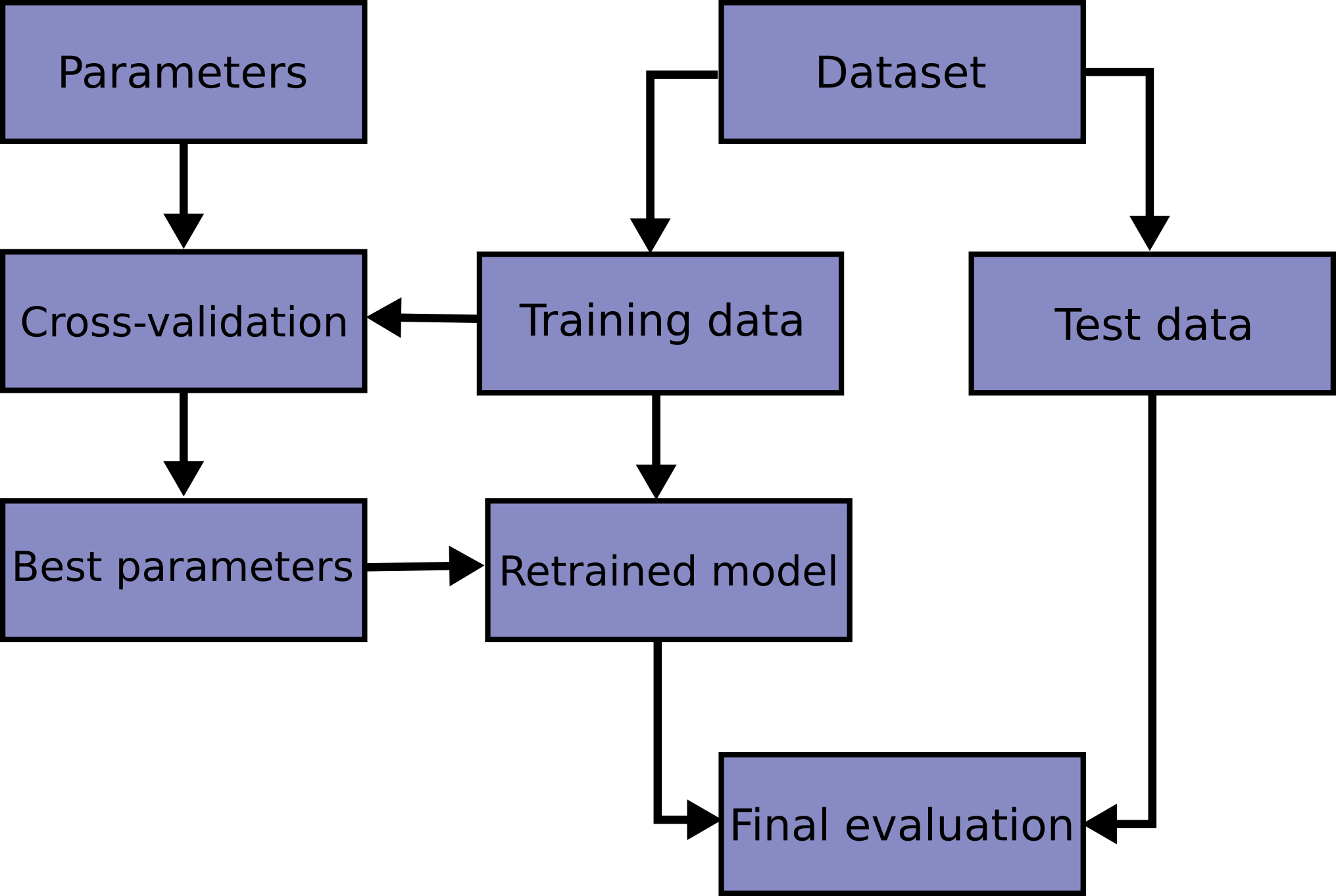
Dividimos el conjunto al inicio, reservando un conjunto de test.
Verificamos los mejores parametros mediante validación cruzada.
Reentrenamos con los mejores parametros y realizamos la evaluación final. (Esto se puede hacer usando GridSearch)
En esta última etapa es donde validamos si existe sobre ajuste. Si existe, se deben incluir parametros para mitigar el sobre ajuste en la validación cruzada y volver al paso 2.
La curva de aprendizaje puede ser necesaria si no logramos mitigar el sobre ajuste, como una forma de evaluar y tener evidencias sobre la deficiencia del tamaño del conjunto de muestras con el que estamos trabajando.

