Laboratorio 5 - Parte 1. Redes recurrentes
Contents
Recuerda que una vez abierto, Da clic en “Copiar en Drive”, de lo contrario no podras almacenar tu progreso
Nota: no olvide ir ejecutando las celdas de código de arriba hacia abajo para que no tenga errores de importación de librerías o por falta de definición de variables.
#configuración del laboratorio
# Ejecuta esta celda!
%load_ext autoreload
%autoreload 2
in_colab = True
import os
if not in_colab:
import sys ; sys.path.append('../commons/utils/'); sys.path.append('../commons/utils/data')
else:
os.system('wget https://raw.githubusercontent.com/jdariasl/ML_2020/master/Labs/commons/utils/general.py -O general.py')
from general import configure_lab5_1
configure_lab5_1()
os.environ['TF_CPP_MIN_LOG_LEVEL'] = '3'
from tensorflow.keras.utils import plot_model
import tensorflow as tf
tf.compat.v1.logging.set_verbosity(tf.compat.v1.logging.ERROR)
from lab5 import *
GRADER, dataset = part_1()
Laboratorio 5 - Parte 1. Redes recurrentes¶
En este laboratorio entrenaremos una Red Neuronal Recurrente para la predicción de una serie de tiempo.
Este problema corresponde a una configuración many-to-one.
En este caso usaremos una serie de tiempo que corresponde al número de pasajeros internacionales por mes, desde el año 1949 hasta el año 1960.
En la siguiente celda visualizamos los datos.
Debemos observar el aparente periodo que existe en nuestra variable. ¿cada cuantos meses parece repertirse el patrón de la serie?
# creamos una variable para
# el tiempo
Time = pd.date_range(np.datetime64('1949-01'), np.datetime64('1961-01'), freq='M')
print("tenemos dispnible nuestra base de datos en el pandas DF 'dataset' \n")
fig, ax = plt.subplots(figsize = (16,6))
ax.plot(Time,dataset)
ax.set_title('International airline passengers')
ax.set_xlabel('Time (months)')
ax.set_xticks( pd.date_range(np.datetime64('1949-01'), np.datetime64('1961-01'), freq='3M'))
plt.xticks(rotation=90)
plt.show()
En nuestro primer ejercicio vamos a explorar, el patrón que observamos en la grafica anterior. Esto tambien nos puede decir que relación existe entre una muestra con las muestras inmediantamente pasadas.
La libreria statsmodel tiene una función que nos sirve para analizar esta relación.
Ejercicio 1 - Exploración del problema¶
Este plot realiza una operación cuyos detalles son explicados en mayor profundidad en esta buena entrada de blog. Pero nuestro laboratorio lo que no interesa entender:
El valor varia entre 1.0 y -1.0.
Cuando el valor de la correlación es 1.0, corresponde el valor maximo indicando una relación positiva entre la variable y su correspondiente lag o retraso.
Cuando el valor de la correlación es -1.0, corresponde el valor mínimo indicando una relación negativa entre la variable y su correspondiente lag o retraso..
0.0 indica que los valores no están relacionados.
el lag indica, el número de retrasos. Si el valor de la correlación en el lag 5 es igual 0.75, indica una relación positiva alta entre el quinto retraso anterior en la mayoria de muestras de nuestra variable de respuesta.
Ahora, grafiquemos la correlación para un maximo de 36 lags de nuestros datos. Esto significa que estamos analizando las relación de una muestras respecto a la 36 muestras pasdas.
Sabiendo que nuestro eje X representa los meses y nuestro eje y representan el numero de pasajeros. Al realizar el analisis de lags estamos determinando si el número de pasajeros de los meses pasados tiene influencia en el nuúmero de pasajeros en el mes acutal.
from statsmodels.graphics import tsaplots
fig, ax = plt.subplots(figsize = (12,7))
# Display the autocorrelation plot of your time series
fig = tsaplots.plot_acf(dataset.passengers, lags=range(1,37), ax = ax)
ax.set_xticks( range(1,37))
ax.scatter(range(0,37,12), 0.1*np.ones(4), c = 'r', marker = "v")
plt.show()
reforzando el entendimiento, observando la grafica anterior:
cuando hay un lag = 5 (es decir evaluar que tan relacionadas estas las 5 muestras anteriores), tenemos una autocorrelación \(\approx\) 0.75
cuando hay un lag = 25 (es decir evaluar que tan relacionadas estas las 25 muestras anteriores), tenemos una autocorrelación \(\approx\) 0.5
Presta atención al patrón que se resalta con las marcas rojas
Vamos observar estas relaciones viendo como los picos de correlación se relacionan con los patrones que vemos. Graficamos los valores de autocorrelación con la grafica de los valores reales.
fig, ax = plt.subplots(figsize = (12,7))
# Display the autocorrelation plot of your time series
fig = tsaplots.plot_acf(dataset.passengers, lags=range(1,37), ax = ax)
ax.set_xticks( range(1,37))
ax.set_ylabel("Valor de correlación")
ax2 = ax.twinx()
ax2.set_ylabel("Numero de pasajeros")
ax2.plot(dataset[0:37], c = 'r')
plt.show()
Ahora bien, para poder aplicar nuestra RNN, debemos transformar nuestros datos. Observa la figura, para entender como debemos transformar los datos.
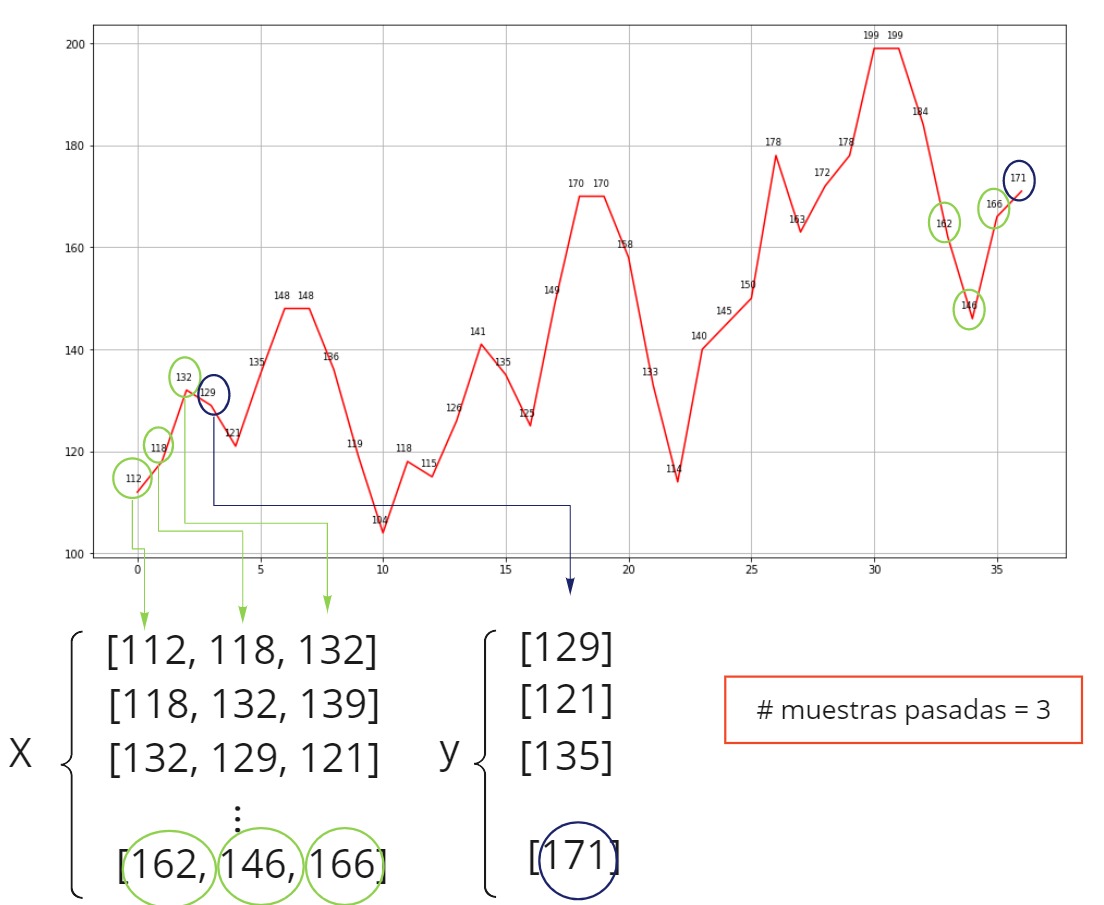
Sabiendo que nuestro anterior analisis de correlación nos indico como están relacionadas las muestras pasadas y al entender que la preparación de datos consiste en usar las muestras pasadas para predicir la muestra siguiente. Responde la siguiente pregunta.
#@title Pregunta Abierta
#@markdown ¿Cual podria ser el numero maximo de muestras pasadas para transformar nuestros conjunto de datos?
respuesta_1 = "" #@param {type:"string"}
Ahora, realicemos el ejercicio de transformar nuestros datos a la forma requerida.
#ejercicio de codigo
def create_dataset(dataset, look_back=1):
"""funcion que crea dataset apto para RNN
dataset: matriz numpy con el conjunto de datos
look_back: numero de retrasos con los cuales queremos construir
las caracteristicas
Retorna:
un numpy array con los valores de X (debe ser una matrix)
un numpy array con los valores de Y
(debe ser un vector columna, el # de renglones debe ser igual de renglones del numpy de X)
"""
dataX, dataY = [], []
for i in ...
...
return np.array(...), np.array(....))
GRADER.run_test("ejercicio1", create_dataset)
# observemos el funcionamiento de nuestra funcion
x_to_see, y_to_see = create_dataset(dataset.values, 3)
display("primeras muestras de x", x_to_see[0:3])
display("primeras muestras de y", y_to_see[0:3])
Ejercicio 2 - Experimentar con RNN¶
En este laboratorio vamos a explorar una libreria muy popular pero un poco más avanzada, para construir redes neuronales llamadas TensorFlow.
En el siguiente ejercicio vamos a crear una función para construir una RNN usando la libreria mencionada. Ssingar como funcion de perdida el valor dfel error medio absoluto
# ejercicio de código
# usar solo estos objetos
# importados
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
from tensorflow.keras.layers import SimpleRNN
import tensorflow as tf
def create_rnn_model(look_back, num_hidden_neurons):
"""funcion que crear modelo que usa mean_absolute_error
como funcion de perdida
RNN con base al número de lags y numero de neuronas
parametros
look_back (int): numero de retrasos a ejecutar
num_hidden_neurons (int): numero neuronas en la capa oculta
"""
# se inicializa el modelo
# podemos asginar un nombre
model = Sequential(name='rnn')
# adicionar una capa RNN
# reemplace los valores
# asigna el nombre de rnn_layer
rnn_layer = SimpleRNN(...,
input_shape = (1,...),
use_bias=True, name = 'rnn_layer')
# en tensorflow debemos adicionar la capa
# al modelo
model.add(...)
# la red termina con una capa Densa de una salida
model.add(Dense(1, name = "dense_layer"))
# remplace la perdida por el parametro correcto
model.compile(loss=..., optimizer='adam')
return(model)
GRADER.run_test("ejercicio2", create_rnn_model)
# observa la diferencias de los modelos al
# cambiar los parametros
# observe los inputs y output
# Los None hacer referencia al numero de muestras de los conjuntos
# de datos
display("1 retraso, 2 neuronas ocultas", plot_model(
create_rnn_model(look_back = 1,num_hidden_neurons = 2),
show_shapes=True))
display("4 retraso, 1 neurona ocultas", plot_model(
create_rnn_model(look_back = 4,num_hidden_neurons = 1),
show_shapes=True))
Con nuestra funcion que crea modelos, vamos experimentar variando los dos parametros:
número de retrasos
número de neuronas en la capa oculta
Otras condiciones:
Vamos a dejar fijo el # de epocas 50.
Usaremos la metrica del score \(R^{2}\). Recordar que solo usamos la implementaciones de sklearn
En la celda de codigo se propone la implementacion para cambiar las dimensiones de nuestras matrices. Este cambio es requerimiento de Tensorflow.
def datas_as_tensorflow(trainX, testX):
# adaptar para compatibilidad con tensorflow
# la libreria necesita tener los cojuntos de datos de la manera
# (# muestras, 1, # de caraceristicas)
trainX = np.reshape(trainX, (trainX.shape[0], 1, trainX.shape[1]))
testX = np.reshape(testX, (testX.shape[0], 1, testX.shape[1]))
return(trainX, testX)
return(tf.convert_to_tensor(trainX), tf.convert_to_tensor(testX))
#ejercicio de código
def experimentar_rnn(data, look_backs, hidden_neurons):
"""funcion que realiza experimentos para evaluar una RNN de elman usando
el error absoluto medio como medida de error
data: pd.Dataframe, dataset a usar
look_back: List[int], lista con los numero de retrasos a evaluar
hidden_neurons: List[int], list con el numero de neuronas en la capa oculta
retorna: pd.Dataframe con las siguientes columnas:
- lags
- neuronas por capas
- error de entrenamiento
- error de prueba
"""
# Normalizar
scaler = MinMaxScaler(feature_range=(0, 1))
dataset = scaler.fit_transform(data)
# realizar el split
train_size = int(len(dataset) * 0.7)
test_size = len(dataset) - train_size
train, test = dataset[0:train_size,:], dataset[train_size:len(dataset),:]
resultados = pd.DataFrame()
idx = 0
for num_hidden_neurons in hidden_neurons:
for look_back in look_backs:
# aplicar la transformacion
trainX, trainY = create_dataset(train, look_back)
testX, testY = create_dataset(test, look_back)
trainX, testX = ...(trainX, testX)
# creemos el modelo
model = create_rnn_model(..., ....)
# entrenemos el modelo
model.fit(x = ..., y = ..., epochs=..., verbose = 0)
# predecimos en los conjuntos
trainYPred = model.predict(trainX)
testYPred = model.predict(testX)
errorPrueba = ...(y_true = ...)
errorEntrenamiento = r2_score(y_true = ...)
resultados.loc[idx,'lags'] = look_back
resultados.loc[idx,'neuronas por capa'] = ...
resultados.loc[idx,'error de entrenamiento'] = ...
resultados.loc[idx,'error de prueba'] = ..
idx+=1
print("termina para", look_back, num_hidden_neurons)
return (resultados)
GRADER.run_test("ejercicio3", experimentar_rnn)
Ahora vamos a ver los resultados del experimentos:
variando los lags dejando las neuronas por capa fijas
variando las neuronas y dejando los retrasos fijos
experimente con diferentes configuraciones. Por la inicialización aleatorias los resultados pueden cambiar. Preste a los patrones que se van presentando y no a los valores exactos.
# observa el comportamiento de los lags
import seaborn as sns
resultadosRNN = experimentar_rnn(dataset, look_backs = [3,9,12,24,30,36], hidden_neurons=[15])
# plot
ax1 = sns.relplot(data= resultadosRNN, x= 'lags', y = 'error de prueba', kind = 'line', aspect = 2)
ax1.fig.suptitle('efecto del # retrasos')
resultadosRNN = experimentar_rnn(dataset, look_backs = [9], hidden_neurons=[5,15,30,60])
ax2 = sns.relplot(data= resultadosRNN, x= 'neuronas por capa', y = 'error de prueba', kind = 'line', aspect = 2)
ax2.fig.suptitle('efecto del # neuronas')
#@title Pregunta Abierta
#@markdown ¿Por qué seguir aumentando los tiempos de retardo no implica siempre una mejora en la predicción del modelo?
respuesta_2 = "" #@param {type:"string"}
#@title Pregunta Abierta
#@markdown ¿Entre el número de retrasos y de neuronas, que parámetro tiene una mayor influencia en el error de prueba?
respuesta_3 = "" #@param {type:"string"}
Ejercicio 3 - Comparación con MLP¶
En este ejercicio vamos a realizar el mismo ejercicio, pero con un MLP. Con esto vamos a comparar los resultados obtenidos con la RNN.
Para ellos vamos a :
variar los retrasos, que corresponden a las neuronas en la capa de entrada
vamos a dejar solo una capa oculta y vamos a variar el número de neuronas en esta capa
el número de epocas también va ser de 50
el valor por defecto sera usado para el resto de parámetros
Usaremos la metrica del score \(R^{2}\). Recordar que solo usamos la implementaciones de sklearn
#@title Pregunta Abierta
#@markdown ¿explique la principal diferencia entre un MLP y una red recurrente? justificar usando usando como contexto el problema que estamos resolviendo.
respuesta_4 = "" #@param {type:"string"}
#ejercicio de código
def experimentar_MLP(data, look_backs, hidden_neurons):
"""funcion que realiza experimentos para evaluar una MLPusando
MAPE como medida de error
data: pd.Dataframe, dataset a usar
look_back: List[int], lista con los numero de retrasos a evaluar
hidden_neurons: List[int], list con el numero de neuronas en la capa oculta
retorna: pd.Dataframe con las siguientes columnas:
- lags
- neuronas por capas
- error de prueba
- tiempo de entrenamiento
"""
# we need to normalize the dataset before
#
scaler = MinMaxScaler(feature_range=(0, 1))
dataset = scaler.fit_transform(data)
# split into train and test sets
train_size = int(len(dataset) * 0.7)
test_size = len(dataset) - train_size
train, test = dataset[0:train_size,:], dataset[train_size:len(dataset),:]
resultados = pd.DataFrame()
idx = 0
for num_hidden_neurons in hidden_neurons:
for look_back in look_backs:
trainX, trainY = create_dataset(train, look_back)
testX, testY = create_dataset(test, look_back)
# define el MLP para Regresion pasando los parametros adecuados
# pasar random_state = 10 para lograr resultados reproducibles
net = MLPRegressor(hidden_layer_sizes= (num_hidden_neurons), ... , random_state = 10)
# Entrenar la red con los datos de entrenamiento
net...
# Evaluar la red con los datos de entrenamiento y test
trainYPred = net....
testYPred = net...
# Calculo de error
errorPrueba = ...(y_true = testY,...)
errorEntrenamiento = ....(y_true = trainY, y_pred=trainYPred)
resultados.loc[idx,'lags'] = ...
resultados.loc[idx,'neuronas por capa'] = ...
resultados.loc[idx,'error de entrenamiento'] = ...
resultados.loc[idx,'error de prueba'] = ...
idx+=1
return (resultados)
GRADER.run_test("ejercicio4", experimentar_MLP)
resultadosMLP = experimentar_MLP(dataset, look_backs = [3,9,12,24,30,36], hidden_neurons=[10,20,30])
# para ver los resultados
# en esta instruccion se va resaltar el mejor
# error
resultadosMLP.style.highlight_min(color = 'green', axis = 0, subset = ['error de prueba'])
Ejercicio 4 - Comparación con LSTM¶
En nuestro ultimo ejercicio, vamos a comparar los resultados obtenidos hasta ahora con una LSTM. Para ellos vamos a usar volver a usar Tensorflow.
#@title Pregunta Abierta
#@markdown ¿por qué una red LSTM puede ser más adecuada para resolver este problema? justifique
respuesta_5 = "" #@param {type:"string"}
Aca creamos el modelo LSTM usando tensorflow:
from tensorflow.keras.layers import LSTM
def create_lstm_model(look_back, num_hidden_neurons):
"""funcion que crear modelo LSTM con base al número de lags y numero de neuronas"""
model = Sequential()
model.add(LSTM(num_hidden_neurons, input_shape=(1, look_back)))
model.add(Dense(1))
model.compile(loss='mean_squared_error', optimizer='adam')
return(model)
Vamos aseguranos de completar el código para lograr:
Epocas = 50
Pasar los parametros el la función
create_tf_modelUsaremos la metrica del score \(R^{2}\). Recordar que solo usamos la implementaciones de sklearn
#ejercicio de código
import tensorflow as tf
def experimentar_LSTM(data, look_backs, hidden_neurons):
"""funcion que realiza experimentos para evaluar una LSTM usando
MAE Error Absoluto medio
data: pd.Dataframe, dataset a usar
look_back: List[int], lista con los numero de retrasos a evaluar
hidden_neurons: List[int], list con el numero de neuronas en la capa oculta
retorna: pd.Dataframe con las siguientes columnas:
- lags
- neuronas por capas
- error de entrenamiento
- error de prueba
"""
# we need to normalize the dataset before
#
scaler = MinMaxScaler(feature_range=(0, 1))
dataset = scaler.fit_transform(data)
# split into train and test sets
train_size = int(len(dataset) * 0.7)
test_size = len(dataset) - train_size
train, test = dataset[0:train_size,:], dataset[train_size:len(dataset),:]
resultados = pd.DataFrame()
idx = 0
for num_hidden_neurons in hidden_neurons:
for look_back in look_backs:
trainX, trainY = create_dataset(train, look_back)
testX, testY = create_dataset(test, look_back)
# adaptar para compatibilidad con tensorflow
trainX, testX = ...(trainX, testX)
# llama la función definida anteriormente
model = ....(look_back, num_hidden_neurons)
# pasa el las epocas requeridas
model.fit(trainX, trainY, ...., verbose=0)
# Evaluar la red con los datos de test y entrenamiento
trainYPred = model....
testYPred = model...
# Calculo de error
errorTest = ....(y_true = ....)
errorTrain = r2_score(y_true = ....)
resultados.loc[idx,'lags'] = ....
resultados.loc[idx,'neuronas por capa'] = ....
resultados.loc[idx,'error de entrenamiento'] = ...
resultados.loc[idx,'error de prueba'] = ...
idx+=1
print("termina para", look_back, num_hidden_neurons)
return (resultados)
# ignorar los prints!
GRADER.run_test("ejercicio5", experimentar_LSTM)
# demora algunos minutos!
resultadosLSTM = experimentar_LSTM(dataset, [3,9,12,24,30,36], hidden_neurons=[5,10,15])
# para ver los resultados
# en esta instruccion se va resaltar el mejor
# error y tiempo de entrenamiento
resultadosLSTM.style.highlight_max(color = 'green', axis = 0, subset = ['error de prueba'])
# comparemos nuevamente con un RNN simple
resultadosRNN = experimentar_rnn(dataset, look_backs = [3,9,12,24,30,36], hidden_neurons=[5,10,15])
# para ver los resultados
# en esta instruccion se va resaltar el mejor
# error y tiempo de entrenamiento
resultadosRNN.style.highlight_max(color = 'green', axis = 0, subset = ['error de prueba'])
GRADER.check_tests()
#@title Integrantes
codigo_integrante_1 ='' #@param {type:"string"}
codigo_integrante_2 = '' #@param {type:"string"}
esta linea de codigo va fallar, es de uso exclusivo de los profesores
GRADER.grade()

